RaTG13: Quanto è Pericoloso un Parente Stretto di SARS-CoV-2?
- Blog Yazısı
Bat-CoV/RaTG13 (o precedentemente Ra4991) è un betacoronavirus simile alla SARS che infetta i pipistrelli del genere Rhinolophus affinis. È stato scoperto nel 2013 negli escrementi di pipistrelli di una vecchia miniera abbandonata nella contea di Mojiang, nello Yunnan, in Cina. RaTG13, che è stato oggetto di ricerca avviata dopo lo scoppio della pandemia COVID-19, è stato dichiarato il parente più stretto di SARS-CoV-2 con una somiglianza nucleotidica del 96,1% nel febbraio 2020. RaTG13 ha perso questo titolo quando Banal 52, scoperto in Laos/Feuang nel 2022, ha mostrato una somiglianza nucleotidica del 96,8%.
La Storia della Scoperta
Nella primavera del 2012, tre minatori nella contea di Mojiang hanno sviluppato una polmonite mortale. Quando la polmonite non ha risposto ai farmaci, i campioni prelevati dai pazienti sono stati inviati all'Istituto di virologia di Wuhan, temendo che si trattasse di una nuova malattia. Sui campioni sono stati eseguiti i test Ebola, Nipah e SARSr-Cov Rp3, quando si è verificata l'epidemia di COVID-19, sui campioni è stato eseguito anche il test SARS-CoV-2 e sono stati ottenuti risultati negativi.
Negli studi del 2012 e del 2015 per indagare sul possibile patogeno della malattia, sono stati prelevati campioni da pipistrelli, roditori e ratti che vivono nella miniera e nelle sue vicinanze. Sono stati isolati 284 alfacoronavirus e 9 betacoronavirus. Un ceppo del virus isolato dal personale è stato trovato nelle feci dei pipistrelli Rhinolophus affinis ed è stato inizialmente chiamato Bat-CoV/Ra4991. Dopo la pandemia di COVID-19, il nome del virus è stato modificato per essere codificato con le iniziali latine (Rhinolophus affinis), alcune lettere del luogo in cui è stato scoperto (Tongguan) e l'anno in cui è stato scoperto (2013): Bat-CoV /RaTG13.
Struttura e Virologia
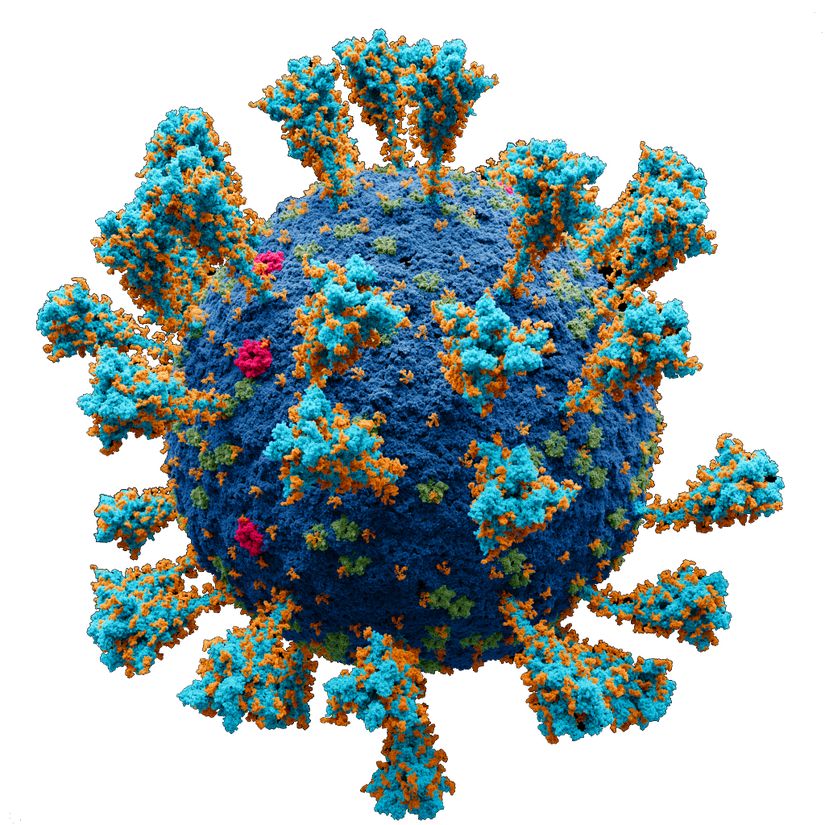
RaTG13 è un virus a RNA a singolo filamento di polarità positiva. Ha una membrana virale a doppio strato lipidico a cui sono fissate le proteine strutturali. Il genoma lungo circa 29.800 nucleotidi codifica per una proteina replicasi, 4 proteine strutturali e 5 proteine accessorie virali. Come tutti i coronavirus, ha 3 tipi di proteine: proteine strutturali, non strutturali e accessorie.
Struttura delle Proteine Strutturali
RaTG13; Contiene quattro diverse proteine: membrana, spike, nucleocapside e involucro:
- Le proteine di membrana codificate dal gene ORF5 sono essenziali per la replicazione virale e conferiscono al virus la sua forma rotonda. È la proteina più abbondante nel genoma ed è lunga circa 230 aminoacidi.
- Le proteine dell'involucro codificate dal gene ORF4 sono incorporate nello strato lipidico e svolgono un ruolo in eventi come la circolazione del materiale intravirale, la formazione di vironi, il germogliamento e sono lunghe 110 aminoacidi.
- Le proteine nucleocapside codificate dal gene ORF9 (o ORF9a) subiscono la massima espressione genica e si legano all'RNA per formare strutture ribonucleoproteiche con una lunghezza media di 220 aminoacidi.
- Le proteine spike codificate dal gene ORF2 si legano ai recettori ACE2, interagendo tra il virus e la cellula ospite e formando l'infezione.
Meccanismo di Infezione
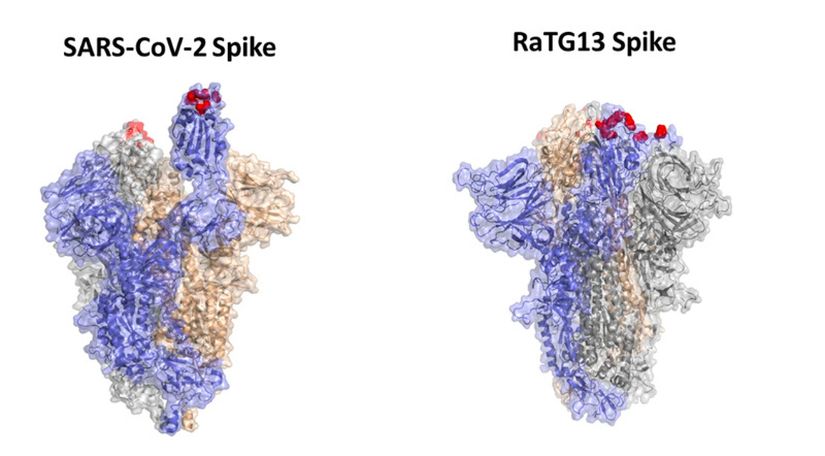
Anche se si presume che i coronavirus simili alla SARS utilizzino l'ACE (enzima di conversione dell'angiotensina) 2, alcuni studi indicano che l'ACE2 per RaTG13 potrebbe non essere funzionale per l'infezione. Fatta eccezione per i pipistrelli, non c'è nessun'altra creatura che RaTG13 sia abbastanza sensibile da causare un'infezione efficace. È meno sensibile agli enzimi ACE2 presenti nelle cellule umane, chiamati hACE2, rispetto a SARS-CoV-2, ma può infettare le cellule umane in casi estremi.
Le proteine della Spike svolgono un ruolo di primo piano per un'infezione efficace nei coronavirus. Ce ne sono 74 in un coronavirus medio e le estensioni del trimero fanno sembrare il virus una corona. Questa proteina consiste di due regioni, S1 e S2, che sono state ben conservate nel processo evolutivo. La regione S1 interagisce con i recettori sulla superficie della cellula ospite durante le prime fasi dell'infezione virale. Questa regione è considerata il sito di legame del recettore (RBD). La regione S2 è responsabile della fusione della membrana tra l'involucro virale e la cellula ospite e consente al genoma virale di entrare nella cellula.
Genetico
Il genoma di RaTG13, contenente circa 29.800 nucleotidi, ha una regione non tradotta chiamata 5'UTR nella regione 5' e una regione non tradotta chiamata 3'UTR nella regione 3'.
Il 5'UTR è responsabile della replicazione virale, della trascrizione e della circolazione intravirale. Ha una struttura secondaria dell'RNA evolutivamente conservata ei primi 150-200 nucleotidi sono altamente strutturati e hanno tre anse staminali conservate (SL1, SL2, SL4). Si stima che il genoma SARS-CoV-2 e RaTG13 sia costituito da 4 anse con uno stelo SL3 aggiuntivo SL1, SL2, SL3 e SL4. È presente anche una struttura più grande, SL5, contenente ORF che esprimono la proteina pp1a.
Il 3'UTR è anche responsabile di importanti attività virologiche come la replicazione virale. Il 3'UTR dei coronavirus simili alla SARS ha una media di 300-500 nucleotidi e ha una struttura secondaria dell'RNA ben conservata.
Geni dell'ORF
ORF1ab (o ORF1a/b) è comune a tutti i coronavirus ed esprime due distinti ORF (ORF1a e ORF1b) nei genomi dei virus simili alla SARS. ORF1ab, noto come gene della replicasi, codifica per le poliproteine non strutturali pp1a e pp1b. Altri geni ORF possono essere elencati come segue:
Evrim Ağacı'nın çalışmalarına Kreosus, Patreon veya YouTube üzerinden maddi destekte bulunarak hem Türkiye'de bilim anlatıcılığının gelişmesine katkı sağlayabilirsiniz, hem de site ve uygulamamızı reklamsız olarak deneyimleyebilirsiniz. Reklamsız deneyim, sitemizin/uygulamamızın çeşitli kısımlarda gösterilen Google reklamlarını ve destek çağrılarını görmediğiniz, %100 reklamsız ve çok daha temiz bir site deneyimi sunmaktadır.
KreosusKreosus'ta her 50₺'lik destek, 1 aylık reklamsız deneyime karşılık geliyor. Bu sayede, tek seferlik destekçilerimiz de, aylık destekçilerimiz de toplam destekleriyle doğru orantılı bir süre boyunca reklamsız deneyim elde edebiliyorlar.
Kreosus destekçilerimizin reklamsız deneyimi, destek olmaya başladıkları anda devreye girmektedir ve ek bir işleme gerek yoktur.
PatreonPatreon destekçilerimiz, destek miktarından bağımsız olarak, Evrim Ağacı'na destek oldukları süre boyunca reklamsız deneyime erişmeyi sürdürebiliyorlar.
Patreon destekçilerimizin Patreon ile ilişkili e-posta hesapları, Evrim Ağacı'ndaki üyelik e-postaları ile birebir aynı olmalıdır. Patreon destekçilerimizin reklamsız deneyiminin devreye girmesi 24 saat alabilmektedir.
YouTubeYouTube destekçilerimizin hepsi otomatik olarak reklamsız deneyime şimdilik erişemiyorlar ve şu anda, YouTube üzerinden her destek seviyesine reklamsız deneyim ayrıcalığını sunamamaktayız. YouTube Destek Sistemi üzerinde sunulan farklı seviyelerin açıklamalarını okuyarak, hangi ayrıcalıklara erişebileceğinizi öğrenebilirsiniz.
Eğer seçtiğiniz seviye reklamsız deneyim ayrıcalığı sunuyorsa, destek olduktan sonra YouTube tarafından gösterilecek olan bağlantıdaki formu doldurarak reklamsız deneyime erişebilirsiniz. YouTube destekçilerimizin reklamsız deneyiminin devreye girmesi, formu doldurduktan sonra 24-72 saat alabilmektedir.
Diğer PlatformlarBu 3 platform haricinde destek olan destekçilerimize ne yazık ki reklamsız deneyim ayrıcalığını sunamamaktayız. Destekleriniz sayesinde sistemlerimizi geliştirmeyi sürdürüyoruz ve umuyoruz bu ayrıcalıkları zamanla genişletebileceğiz.
Giriş yapmayı unutmayın!Reklamsız deneyim için, maddi desteğiniz ile ilişkilendirilmiş olan Evrim Ağacı hesabınıza üye girişi yapmanız gerekmektedir. Giriş yapmadığınız takdirde reklamları görmeye devam edeceksinizdir.
- ORF3a è un gene trovato nei coronavirus simili alla SARS. Sebbene incerto, si pensa che codifichi una proteina long helper di 275 aminoacidi coinvolta nella formazione della viroporina.
- ORF3b si trova anche solo nei virus simili alla SARS e codifica per una breve proteina non strutturale.
- Anche ORF3c è stato scoperto per la prima volta con SARS-CoV-2. Ma in seguito è stato scoperto anche in RaTG13 e altri SARSr-CoV. Codifica una proteina non strutturale lunga 41 aminoacidi di funzione sconosciuta.
- ORF3d, d'altra parte, è un gene che si trova solo nei coronavirus associati a SARS-CoV-2 e RaTG13 piuttosto che in tutti i coronavirus simili alla SARS.È lungo 57 codoni e codifica una proteina non strutturale lunga 57 amminoacidi di funzione sconosciuta .
- ORF6 codifica una proteina helper con una lunghezza media di 60 aminoacidi nei virus associati alla SARS, che varia da virus a virus.
- ORF7a codifica per le proteine transmembrana che si pensa siano immunomodulazione e antagonismo dell'interferone, sebbene la sua funzione in RaTG13 e SARS-CoV-2 sia incerta. Anche ORF7b, che si sovrappone a ORF7a, è un gene che codifica per una proteina helper di funzione sconosciuta.
- ORF8, che è simile a ORF7a nella struttura, codifica per la proteina helper virale denominata NS8. Questa proteina è una proteina con un dominio immunoglobulinico la cui funzione e meccanismo sono sconosciuti, che interagisce con il sistema di difesa della cellula ospite in SARS-CoV-2 e RaTG13.
- ORF9b, precedentemente noto come ORF13, è il gene che codifica per una proteina virale regolatrice e accessoria con una lunghezza media di 90-100 aminoacidi nei coronavirus simili alla SARS.
- ORF9b è un gene che si sovrappone alla proteina ORF9 che codifica per la proteina Nucleocapsid.
- ORF9c (precedentemente ORF14) è un gene con una lunghezza media del codone di 70-73 in SARS-CoV-2 e RaTG13. I dati bioinformatici indicano che ORF9c potrebbe non essere un gene che codifica una proteina funzionale.
- ORF10 è un gene lungo 38 codoni e codifica per una proteina accessoria evolutivamente non conservata. Come con ORF9c, i dati bioinformatici indicano che ORF10 potrebbe non essere un gene che codifica per una proteina virale regolatrice.
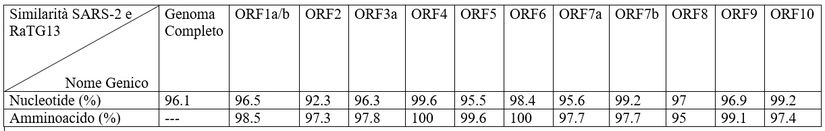
Confronto del Genoma
Il rapporto di somiglianza genetica tra RaTG13 e SARS-CoV-2 è mostrato nella tabella sottostante.
Evoluzione, Tassonomia e Filogenetica
In termini tassonomici, RaTG13 appartiene alla classe dei Sarbecovirus, che fa parte della sottofamiglia dei Betacoronavirus della famiglia dei Coronavirus, scientificamente chiamata Orthocoronavirinae. Appartiene al clade dei virus associati a SARS-CoV-2 all'interno di Sarbecovirus.
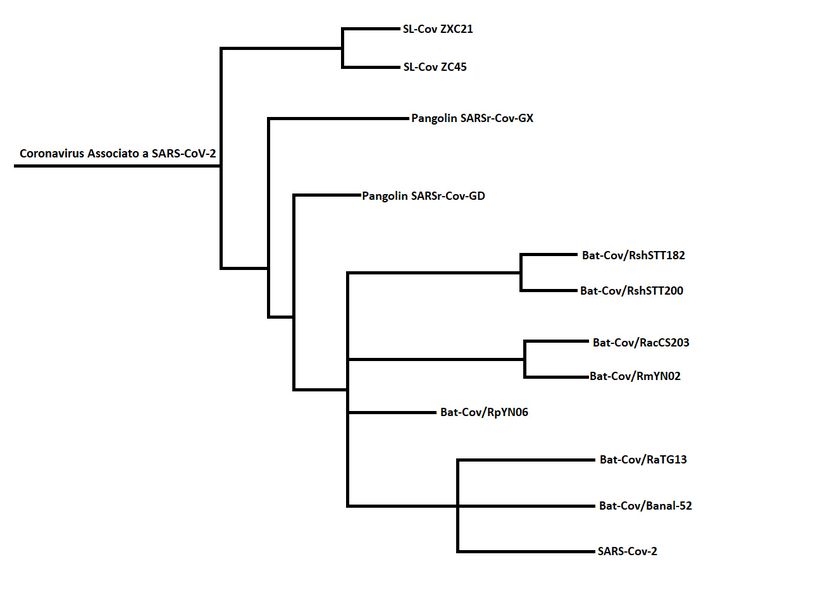
L'evoluzione del RaTG13
Non esiste una relazione ancestrale-discendente tra SARS-CoV-2 e RaTG13 (cioè uno non si è evoluto direttamente dall'altro). L'evoluzione di entrambi i virus non è ancora chiara; tuttavia, una caratteristica distintiva dei coronavirus simili alla SARS è la tendenza alla ricombinazione genetica con altri coronavirus nell'organismo ospite che infettano. L'evoluzione dei SARSr-CoV, che si è discostata e si è evoluta molto presto dal resto dei betacoronavirus, è unica. Mentre questi passi evolutivi sono prevedibili, non sono chiari per RaTG13 e SARS-CoV-2. Sono in fase di studio questioni come quando si è evoluto l'antenato comune dei due virus e l'ospite intermedio di SARS-CoV-2. Un metodo di misurazione prevede che la divergenza dei due virus dal loro antenato comune possa variare da 18 a 71,4 anni. Secondo questa previsione, è prevista anche l'esistenza di altri SARSr-CoV che sono più simili a RaTG13 che a SARS-CoV-2 e che si sono discostati dall'antenato comune in un periodo precedente.
- 1
- 1
- 0
- 0
- 0
- 0
- 0
- 0
- 0
- 0
- 0
- 0
Evrim Ağacı'na her ay sadece 1 kahve ısmarlayarak destek olmak ister misiniz?
Şu iki siteden birini kullanarak şimdi destek olabilirsiniz:
kreosus.com/evrimagaci | patreon.com/evrimagaci
Çıktı Bilgisi: Bu sayfa, Evrim Ağacı yazdırma aracı kullanılarak 26/04/2025 02:49:24 tarihinde oluşturulmuştur. Evrim Ağacı'ndaki içeriklerin tamamı, birden fazla editör tarafından, durmaksızın elden geçirilmekte, güncellenmekte ve geliştirilmektedir. Dolayısıyla bu çıktının alındığı tarihten sonra yapılan güncellemeleri görmek ve bu içeriğin en güncel halini okumak için lütfen şu adrese gidiniz: https://evrimagaci.org/s/14678
İçerik Kullanım İzinleri: Evrim Ağacı'ndaki yazılı içerikler orijinallerine hiçbir şekilde dokunulmadığı müddetçe izin alınmaksızın paylaşılabilir, kopyalanabilir, yapıştırılabilir, çoğaltılabilir, basılabilir, dağıtılabilir, yayılabilir, alıntılanabilir. Ancak bu içeriklerin hiçbiri izin alınmaksızın değiştirilemez ve değiştirilmiş halleri Evrim Ağacı'na aitmiş gibi sunulamaz. Benzer şekilde, içeriklerin hiçbiri, söz konusu içeriğin açıkça belirtilmiş yazarlarından ve Evrim Ağacı'ndan başkasına aitmiş gibi sunulamaz. Bu sayfa izin alınmaksızın düzenlenemez, Evrim Ağacı logosu, yazar/editör bilgileri ve içeriğin diğer kısımları izin alınmaksızın değiştirilemez veya kaldırılamaz.








